今日研读
PRIME:一种可提高NSCLC进展风险预测能力的人工智能模型
研究背景►
尽管循环肿瘤DNA(ctDNA)微小残留病(MRD)在肿瘤预后预测中展现出重要价值,但对于早期或局限性
研究方法►
本研究从6个队列中收集了接受手术或根治性放化疗的I-III期NSCLC患者的液体活检数据、基于血液的基因组改变、临床病理特征和生存结局。PRIME(基于ctDNA-MRD、突变及临床治疗特征的可解释机器学习进展风险预测模型)通过4个队列中的6种机器学习算法进行训练,并在2个独立队列中进行验证。采用曲线下面积(AUC)评估模型性能,通过SHapley加法解释(SHAP)对模型进行解释。此外,利用来自癌症基因组图谱的430例II-III期NSCLC患者肿瘤组织的全外显子测序(WES)或全基因组测序(WGS)数据以及1149例受试者的RNA测序(RNA-seq)数据,旨在验证外周血中检出的基因突变的预后价值,并深入探究其潜在作用机制。
研究结果►
本研究对包含来自493例患者的781份血液样本的全球数据集进行了分析。结果显示,临床分期、治疗前ctDNA、治疗后MRD、血液中Kelch样ECH相关蛋白1(KEAP1)、
WES/WGS和RNA-seq证实了KEAP1、STK11和CDKN2A突变的不良预后效应,其特征为肿瘤微环境受抑制和体液免疫减弱。
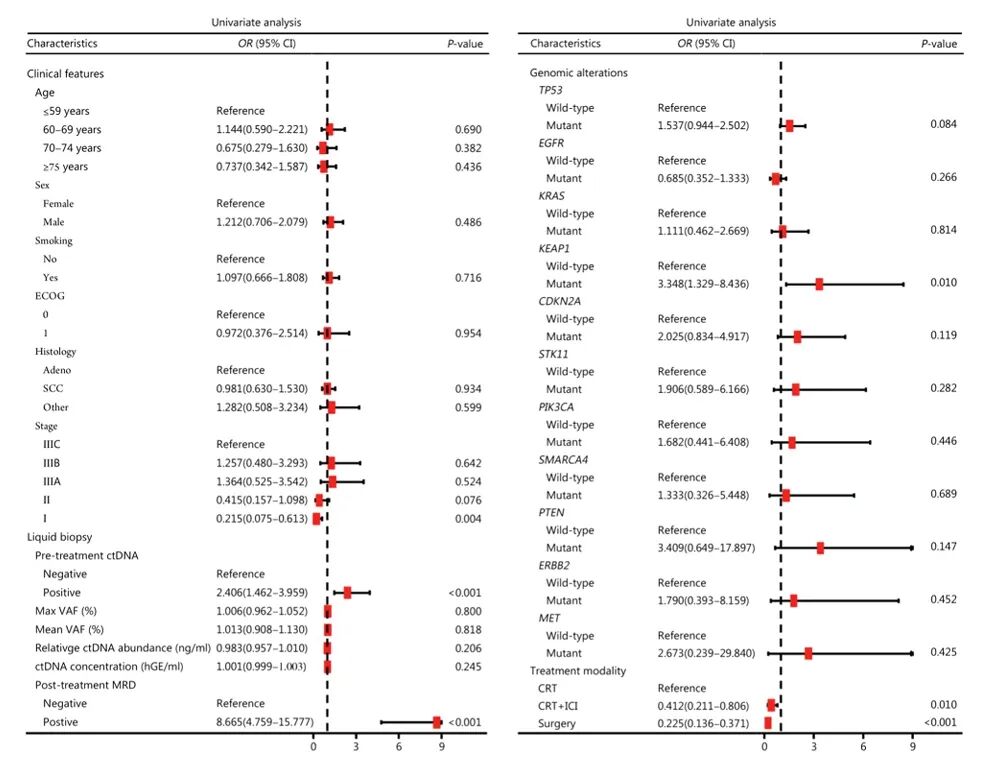
训练集中疾病进展潜在预测因素的森林图(n = 345)
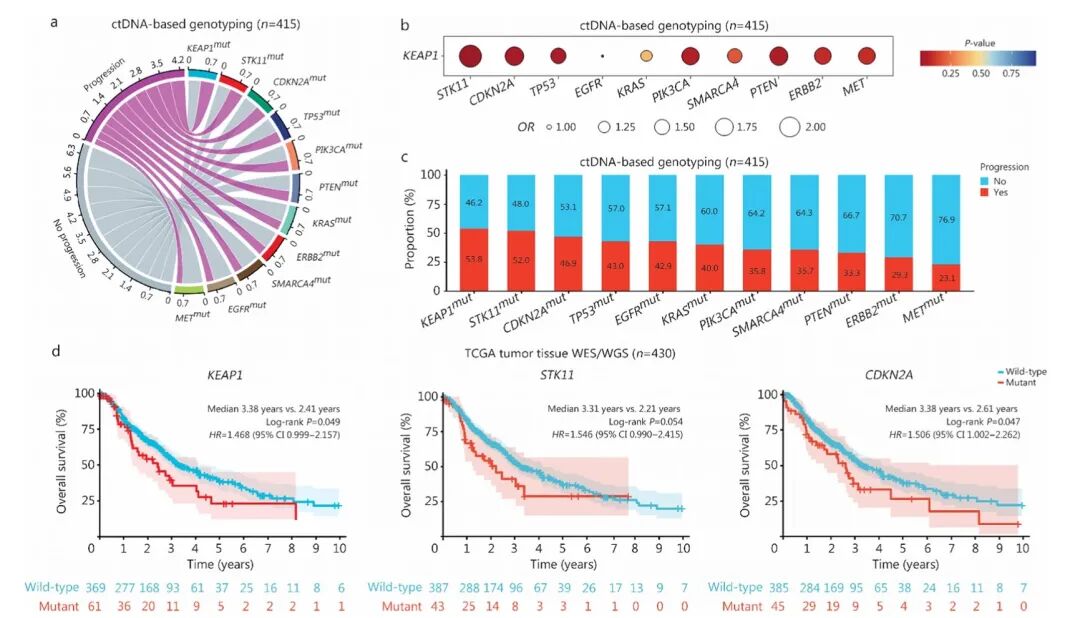
用于疾病进展的基因组突变生物标志物
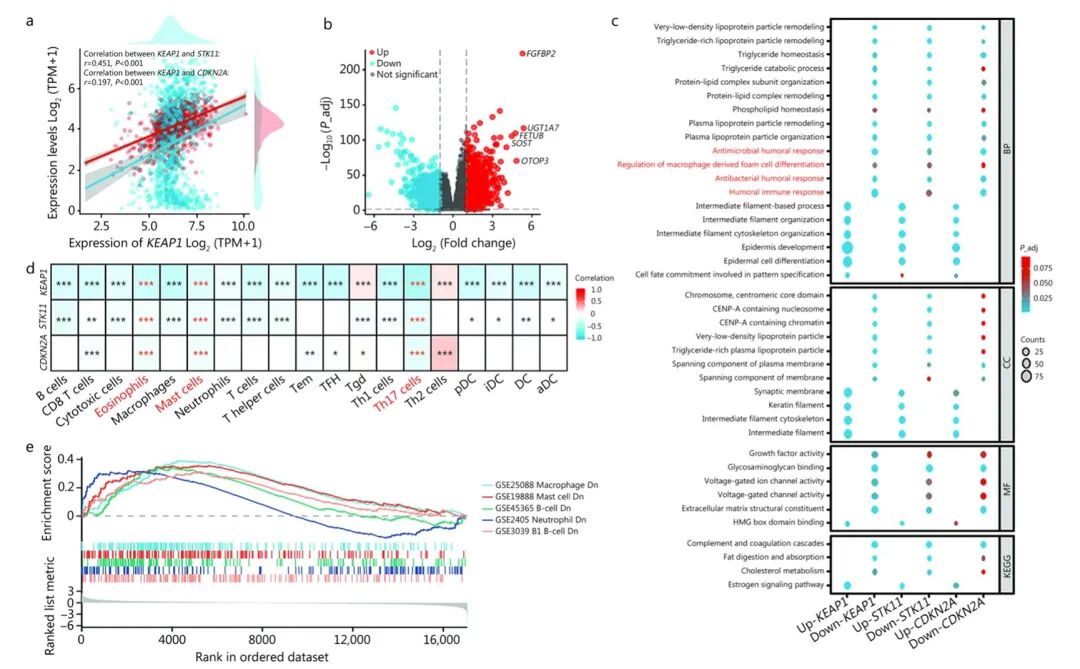
KEAP1-、STK11-和CDKN2A-突变肿瘤的免疫景观
神经网络(NN)模型在训练集(AUC=0.85,95% CI:0.81-0.89)和验证集(AUC=0.82,95% CI:0.74-0.89)中对治疗失败风险的预测表现最优。SHAP分析表明,MRD(+0.306)、治疗方式(+0.128)和治疗前ctDNA(+0.043)位列贡献度前三名。NN-PRIME的表现优于单一的液体活检生物标志物和临床治疗特征,并且在不同的临床场景中都展现出稳定的稳健性。
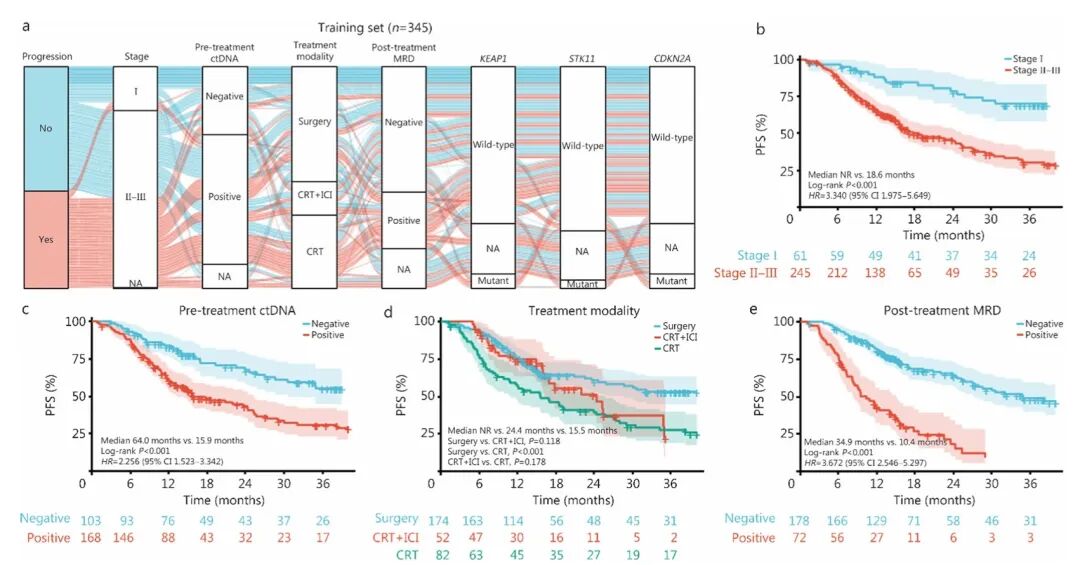
训练集中已识别的多模态特征的预测效果(n = 345)
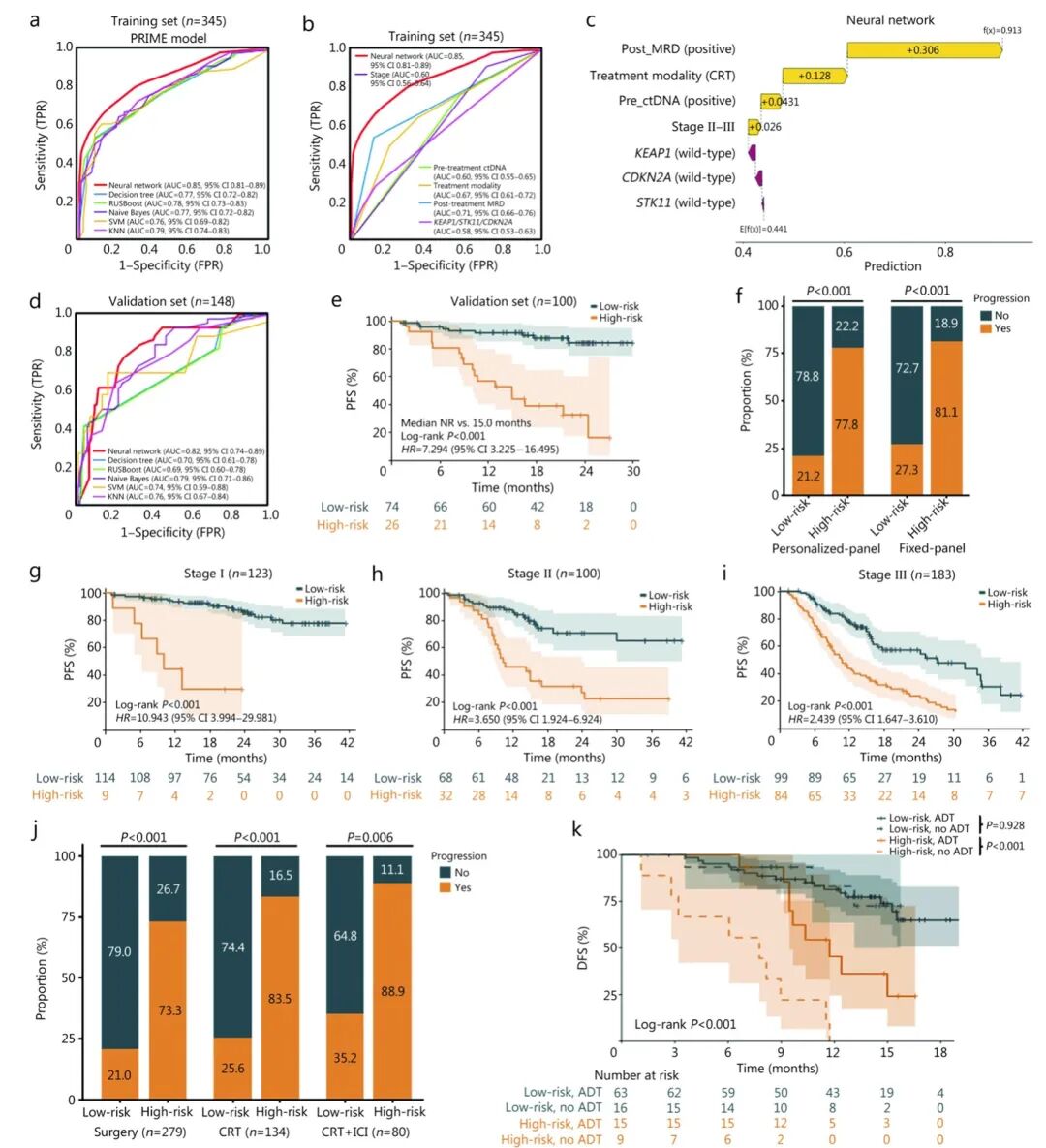
基于NN的PRIME模型构建、解读与验证
研究结论►
作为一种整合了易于获取且关键的临床基因组预测因子的可解释模型,PRIME实现了性能的提升,能够进行早期结局预测、精细化风险分层,为个体化临床决策提供了可靠依据。
参考文献:Wang Y, Xiang YB, et al. PRIME: an interpretable artificial intelligence model based on liquid biopsy improves prediction of progression risk in non-small cell lung cancer. Mil Med Res. 2026 Jan 6;12(1):94.
编辑:Kenken
审校:Faline
排版:Kenken
执行:Faline
医脉通是专业的在线医生平台,“感知世界医学
本平台旨在为医疗卫生专业人士传递更多医学信息。本平台发布的内容,不能以任何方式取代专业的医疗指导,也不应被视为诊疗建议。如该等信息被用于了解医学信息以外的目的,本平台不承担相关责任。本平台对发布的内容,并不代表同意其描述和观点。若涉及版权问题,烦请权利人与我们联系,我们将尽快处理。

(本网站所有内容,凡注明来源为“医脉通”,版权均归医脉通所有,未经授权,任何媒体、网站或个人不得转载,否则将追究法律责任,授权转载时须注明“来源:医脉通”。本网注明来源为其他媒体的内容为转载,转载仅作观点分享,版权归原作者所有,如有侵犯版权,请及时联系我们。)